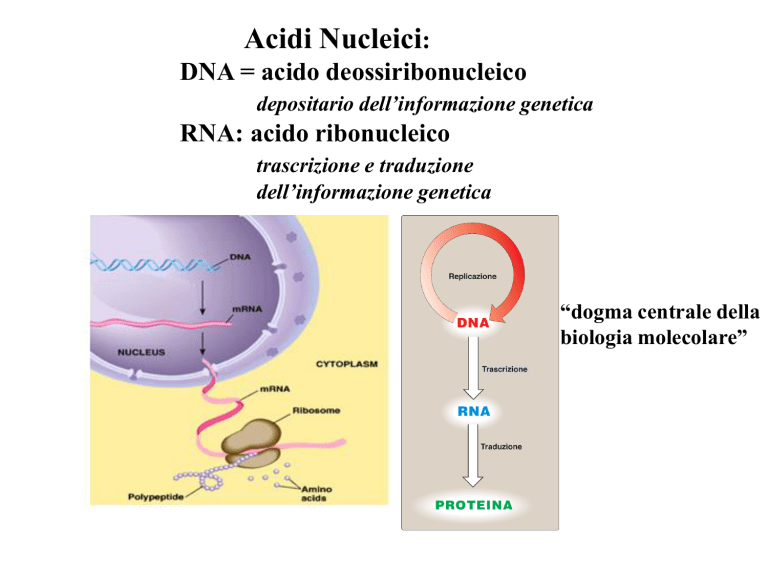
Acidi Nucleici:
DNA = acido deossiribonucleico
depositario dell’informazione genetica
RNA: acido ribonucleico
trascrizione e traduzione
dell’informazione genetica
“dogma centrale della
biologia molecolare”
In alcuni virus l’informazione
genetica è codificata nel RNA
La trascrittasi inversa
converte l’RNA virale
in DNA e ne permette
l’integrazione nel genoma
della cellula ospite
Unità costitutive degli acidi nucleici: nucleotidi
Base
azotata
DNA
RNA
Citosina
Timina
Citosina
Uracile
DNA
RNA
Adenina
Guanina
Adenina
Guanina
RNA
DNA
Base azotata + zucchero =
NUCLEOSIDE
H2O
Base azotata + zucchero + fosfato =
NUCLEOTIDE
Nucleoside Monofosfato
(deossinucleotidi)
(nucleotidi)
Base azotata + zucchero + fosfato =
NUCLEOTIDE
Nucleoside Monofosfato
Nucleoside Difosfato
Nucleoside Trifosfato
Tutti i nucleotidi assorbono luce ultravioletta
Nucleotidi
(acidi nucleici)
Amminoacidi aromatici
(proteine)
Spettri di assorbimento
Gli acidi nucleici sono polimeri
lineari di nucleotidi
Oligonucleotidi…
Polinucleotidi…
Struttura secondaria
DNA
RNA
Il DNA ha una struttura a doppio filamento
l’RNA ha una struttura a filamento singolo
Appaiamento complementare delle basi:
A-T (2 legami H)
C-G (3 legami H)
Interazioni di van der
Waals tra coppie di
basi sovrapposte
(impilamento – stacking)
Per ogni molecola di DNA il contenuto di A è uguale al contenuto di T
e il contenuto di C è uguale al contenuto di G (regola di Chargaff)
Appaiamento complementare delle basi:
A-T (2 legami H)
C-G (3 legami H)
Conseguenza della complementarietà delle basi…
l’informazione contenuta nella sequenza di un
filamento è conservata nella sequenza dell’altro
…ATGCTAACC….
…TACGATTGG….
Eliche destrorse
bp = base pair
Kb = migliaia di coppie di basi
Elica sinistrorsa
(favorita da alternanza
purine-pirimidine,
es: pCGCGCG….)
bp/giro
10.5
Diametro (Å)
20
Inclinazione basi 6°
11
26
20°
solco maggiore
più profondo
solco minore
meno profondo
12
18
7°
solco maggiore poco
profondo
solco minore
stretto e profondo
Il DNA a doppia elica può essere denaturato
La denaturazione del DNA
è un processo reversibile
La riassociazione dei filamenti
“annealing” è rapida e avviene
in una sola tappa se la separazione
è incompleta
Se i filamenti sono completamente
separati la riassociazione avviene
in due tappe: 1) appaiamento di
un breve segmento complementare
(fase più lenta); 2) rinaturazione
completa (fase rapida)
La denaturazione si accompagna ad un
aumento dell’assorbanza a 260 nm:
effetto ipercromico
a
Replicazione del DNA
Enzimi coinvolti:
DNA polimerasi
DNA ligasi
elicasi
topoisomerasi
-Semiconservativa
-Bidirezionale
-Semidiscontinua
Replicazione semiconservativa: Proposta da Watson e Crick - 1953
La replicazione del DNA
è semiconservativa:
Ciascuna molecola figlia
È costituita da una catena
Parentale e una catena
neosintetizzata
Esperimento di Meselson-Stahl
Replicazione
semiconservativa
Nei procarioti la replicazione del DNA inizia in un unico sito
(origine della replicazione)
Negli eucarioti la replicazione inizia in più siti
Sia nei procarioti che negli eucarioti la replicazione è bidirezionale
La sintesi delle nuove molecole di DNA è catalizzata da
DNA polimerasi
-Stampo
-3’-OH libero (innesco/catena in allungamento)
-Nucleosidi 5’-trifosfato (tutti e quattro i nucleotidi
devono essere presenti)
-Mg2+
Quante DNA polimerasi?....replicazione
ricombinazione
riparazione
Processività = numero di
nucleotidi aggiunti prima
che la polimerasi si
dissoci
Velocità di replicazione
in E. coli =
circa 1000 nucleotidi/sec
Enzima principale della replicazione
riparazione
Soluzione al problema 1: -sintesi continua nella direzione di avanzamento
della forcella di replicazione (filamento guida/catena veloce)
-Sintesi discontinua nella direzione di allontanamento dalla forcella di
replicazione (catena lenta: frammenti di Okazaki)
La replicazione del DNA è semidiscontinua
Problema 2: chi fornisce l’innesco?...
Soluzione:
DNA primasi: è una RNA polimerasi che
sintetizza un breve tratto di RNA (circa 10
nucleotidi) complementare allo stampo di
DNA (RNA primer).
La DNA primasi utilizza ribonucleosidi 5’trifosfato e catalizza la formazione del
legame fosfodiestere liberando PPi
La DNA polimerasi aggiunge poi
deossiribonucleotidi all’estremità 3’-OH
del RNA primer
La DNA polimerasi utilizza deossiribonucleotidi
5’-trifosfato e catalizza la formazione del
legame fosfodiestereo liberando PPi
Rimozione del primer e
sostituzione con un tratto
di DNA da parte della DNA
polimerasi I
Saldatura dei frammenti di Okazaki
DNA LIGASI
Reazione a più passggi
1. Attivazione del gruppo fosforico 5’ al
punto di interruzione
2. Formazione del legame fosfodiestereo
Negli Eucarioti la replicazione del DNA avviene durante la fase S
del ciclo cellulare
La trascrizione è molto simile alla replicazione
per quanto riguarda:
Meccanismo chimico
Direzione di sintesi
richiesta di uno stampo
La trascrizione differisce dalla replicazione perché:
Non richiede un primer
Interessa solo brevi segmenti della molecola di DNA
Nel tratto trascritto, uno solo dei filamenti funge da stampo
L’inizio della trascrizione avviene a livello di sequenze
specifiche(regione del promotore) riconosciute dalla RNA
polimerasi
Funzione del promotore:
indica l’inizio del gene che deve essere trascritto
La trascrizione produce più copie
dello stesso gene
Le topoisomerasi rimuovono
i superavvolgimenti
L’RNA polimerasi non ha attività esonucleasica e quindi non può
effettuare “proofreading”
Frequenza di errore: 1 ogni 104-105 ribonucleotidi incorporati nell’RNA
Elevata processività
Come avviene la trascrizione negli Eucarioti?....
Negli Eucarioti sono presenti TRE tipi di RNA polimerasi
RNA polimerasi I (Pol I)
sintesi di RNA pre-ribosomiale
(un unico trascritto contenente
i precursori di rRNA 18S, 5.8S, 28S
RNA polimerasi II (Pol II)
sintesi degli mRNA
RNA polimerasi III (Pol III)
sintesi dei tRNA, rRNA 5S
piccoli RNA
promotore
Fattori di trascrizione (TF)
proteine che regolano
la trascrizione,
ma non sono subunità
della RNA polimerasi
di inizio
Modificazioni post-trascrizionali degli mRNA eucariotici
(maturazione degli mRNA)
Aggiunto prima che la
sintesi del trascritto
primario sia completata
Introni = sequenze non codificanti (eliminati con lo splicing)
Esoni = sequenze codificanti
-guanililtransferasi (nucleo) – da GTP
-guanina 7-metiltransferasi (citoplasma)
cappuccio 5’:
- Protegge l’mRNA dalle ribonucleasi
- Viene riconosciuta da proteine che
legano il ribosoma
Coda di poliA (40-200 nucleotidi)
Aggiunta dalla poliadenilato
polimerasi (nucleo)
Stabilizza l’mRNA
Ne favorisce l’uscita dal nucleo
SPLICING: eliminazione degli introni
snRNP = piccole particelle
ribonucleoproteiche nucleari
(proteine + snRNA)
Splicing alternativo
TRADUZIONE
Il linguaggio a 4 lettere (basi) del
DNA/RNA viene tradotto nel
linguaggio a 20 lettere (amminoacidi)
delle proteine
Codone = tripletta di nucleotidi codificante
per un amminoacido
Caratteristiche del codice genetico:
•Codice a triplette
•Ridondante (o degenerato)
(codoni sinonimi)
•Specifico (non ambiguo)
•Universale
•Non sovrapposto
•Privo di punteggiatura
Il tRNA ha la funzione di “traduttore”(o adattatore)
Per ogni amminoacido c’è almeno un tRNA specifico
Negli Eucarioti sono presenti circa 50 specie di tRNA
Il riconoscimento codone-anticodone
avviene secondo le regole dell’appaiamento
complementare e antiparallelo
Le prime due basi dell’anticodone formano
sempre appaiamenti stabili, mentre la terza
base forma appaiamenti più deboli (base
oscillante, wobble).
Conseguenza funzionale: un dato tRNA può
riconoscere più di un codone
(Ala)
Ansa TyC
Ansa D
Ansa
variabile
Ansa
Dell’anticodone
= pseudouridina
I = inosina
T= ribotimidina
D = 5,6-diidrouridina
m1I= 1-metilinosina
m1G= 1-metilguanosina
m2G= N2-dimetilguanosina
Caratteristiche strutturali del t-RNA
-Lunghezza: 73-93 nucleotidi
-Residuo pG all’estremità 5’ (nella
maggior parte dei t-RNA)
-Sequenza CCA all’estremità 3’
-Struttura secondaria a trifoglio
-Presenza di basi modificate nelle
regioni non appaiate
-Struttura tridimensinale a “L” ribaltata
Step 1: attivazione dell’amminoacido
+ H2O
2Pi
Step 2: formazione dell’amminoacil-tRNA
Il legame tra il t-RNA e l’amminoacido corrispondente
è catalizzato dalle amminoacil-tRNA sintetasi
Per ogni coppia tRNA-amminoacido esiste una
amminoacil-tRNA sintetasi specifica
Le amminoacil-tRNA sintetasi hanno attività di
“correzione delle bozze”
Che cosa determina il corretto posizionamento del mRNA sui ribosomi?...
Nei Procarioti: Sequenza di Shine-Dalgarno
Negli Eucarioti :
la subunità 40S si lega al cappuccio presente all’estremità 5’
dell’mRNA e poi si sposta fino ad incontrare il codone di inizio.
PAB = proteina che lega
il poli-A
eIF = eukaryotic Initiation
Factor
Tappe della sintesi proteica
1. Inizio
2. Allungamento
3. Terminazione
Assemblaggio dei componenti
del sistema di traduzione
IF = fattori di inizio
P= sito peptidilico
A = sito amminoacilico
Tappe della sintesi proteica
1. Inizio
2. Allungamento
3. Terminazione
Assemblaggio dei componenti
del sistema di traduzione
Nei procarioti il primo amminoacido
è formil-Metionina, trasportato dal
formil-Met-tRNA
Negli eucarioti la sintesi comincia
con Met.
Solo il fMet-tRNA si lega
prima al sito P
Tutti gli altri amminoacil-tRNA
si posizionano prima nel sito A
e solo successivamente nel sito
P e nel sito E
Tappe della sintesi proteica
1. Inizio
2. Allungamento
3. Terminazione
Assemblaggio dei componenti
del sistema di traduzione
E= sito di uscita (tRNA scarichi)
P= sito peptidilico
A = sito amminoacilico
Sia la subunità minore che la subunità
maggiore contribuiscono alla formazione
dei siti P e A
Il sito E è localizzato completamente sulla
subunità maggiore
COMPLESSO DI INIZIO
Tappe della sintesi proteica
1. Inizio
2. Allungamento
3. Terminazione
Posizionamento del secondo
amminoacil-tRNA nel sito A
Formazione del legame
peptidico
Traslocazione
Tappe della sintesi proteica
1. Inizio
2. Allungamento
3. Terminazione
Formazione del legame
peptidico
catalizzata dall’attività
peptidil-transferasica
dell’ RNA 23S (ribozima)
Tappe della sintesi proteica
1. Inizio
2. Allungamento
3. Terminazione
traslocazione
Tappe della sintesi proteica
1. Inizio
2. Allungamento
3. Terminazione
RF =
Fattori di rilascio
Si libera la proteina completa
I ribosomi, l’mRNA, i tRNA, i vari
fattori proteici possono essere
riutilizzati nella sintesi di un
altro polipeptide
Una molecola di mRNA può essere tradotto simultaneamente
da numerosi ribosomi (polisomi)
La catena polipeptidica comincia a ripiegarsi già durante la sintesi
Può andare incontro a modificazioni dopo il completamento della sintesi
La proteina finale – ripiegata nella sua conformazione nativa – è
biologicamente attiva












