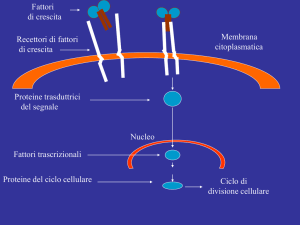
Fattori
di crescita
Membrana
citoplasmatica
Recettori di fattori
di crescita
Proteine trasduttrici
del segnale
Nucleo
Fattori trascrizionali
Proteine del ciclo cellulare
Ciclo di divisione cellulare
I secondi messaggeri
tirosinchinasi
GAP
PLC
adattatori
Le Proteine G
Receptor associated binding proteins
c-ras family
Le Proteine G
MONOMERICHE
Ras, Rho, Rab, Arf, Ran
ETEROTRIMERICHE
GDP
α
β
γ
Le proteine G monomeriche (e la subunità Gα delle
eterotrimeriche) “ciclano” tra le forme che legano
GDP o GTP
GDP
inattiva
GTP
attiva
Effettore
Ras (p21ras) è una piccola (21 kDa) proteina monomerica che lega il GTP o il
GDP e ha un’attività GTPasica intrinseca
Il fattore di scambio
del nucleotide
guanidinico interagisce
con ras
p21ras
GTP
INATTIVA
p21ras
ras attivata interagisce
con il componente
successivo della catena
di signalling e l’attiva
GDP
GDP
GTP
p21ras
ATTIVA
Pi
Ras GTPasi stimolata
dall’associazione con
la GTPase-activating
protein (GAP)
p21ras
GDP
L’attività GTPasica
intrinseca idrolizza il
GTP a GDP e Pi
Alcune proteine legano
e aiutano le GTP:
GAPs, GTPase
Activating Proteins,
promuovono l’idrolisi del
GTP.
GAP may provide an essential active site residue,
and/or promote a conformation that favors catalysis.
Le RGS proteins, which are negative regulators of
G protein signaling, function as GAPs to stimulate
GTP hydrolysis by Gα.
GEFs, Guanine Nucleotide Exchange Factors,
promote GDP/GTP exchange.
The activated receptor (GPCR) serves as GEF for a
heterotrimeric G protein.
insulina
GTP
SOS
GRB2
Ras
attivo
P
P
P
P
Recettore
Recettore inattivo
attivo
Ras attivato attiva la via MAPK attraverso la chinasi Raf
GTP
Raf attivo e
localizzato
sulla
membrana
ATP
Ras
Pi
Raf
ADP
MEK
Raf inattivo
e citosolico
Raf
ERK
Attivazione di
fattori di
trascrizione
ligandodipendenti
Ras attivato attiva la via MAPK attraverso la MAPKKK Raf
ras
GTP
ras
GTP
Raf attivo e localizzato sulla membrana
Raf inattivo e citosolico legato alle proteine 14-3-3
Ras è un Oncogene
p21ras comprende quattro proteine correlate: H-Ras (Harvey),
K-Ras (Ka e Kb-, Kirsten) e N-ras (neuronal)
p21ras è associato alla membrana tramite
farnesilazione di un amminoacido ciascuna isoforma differisce
pricipalmente nel C-terminale da modificazioni posttranscrizionali.
miristico
Gαs
farnesilico
palmitico
ras
Gα
βγ
βγ
farnesilico
Gα
Geranil-geranilico
Gα
βγ
a tre delle quattro isoforme è aggiunto un gruppo palmitoilico Mutazioni
attivanti di p21ras lo bloccano nello stato GTP-legato
βγ
Ras
Gli oncogeni ras sono generati nel tumore da mutazioni in seguito ad
esposizione ad agenti carcinogeni
La mutazione puntiforme con conseguente sostituzione della valina con una
glicina causa soltanto una leggera alterazione nella struttura tridimensionale
di Ras.
Sono state anche riscontrate altre sostituzioni di amminoacidi in posizione
13 e 61, negli oncogeni ras nei tumori umani.
Oltre alla mutazione dominante nel gene ras, anche una mutazione
recessiva di perdita di funzione nel gene NF1 conduce all'attivazione
costitutiva di Ras.
NF1 codifica per GAP.
Gene
Funzione
Tumori associati a
mutazioni somatiche
NF-1
Inibizione di ras
Shwannomi
Tumori associati
a mutazioni ereditarie
Neurofibromatosi tipo 1
Gli individui con neurofibromatosi hanno ereditato un singolo allele del mutante NF1; la
mutazione somatica successiva nell'altro allele conduce a formazione dei neurofibromi,
tumori benigni delle cellule che circondano i nervi.
Recettore attivato
tirosin chinasi
Ras
P-Y
Y-P
P-Y
Y-P
Sos
GDP
Shc
Grb2
GTP
NF-1 è una GAP
(GTPase Activating Protein)
Ras
Gap
Raf
GTP
NF1
Ras
GDP
GDP
Forma inattiva
Segnale a valle
Forma attiva
Ras
Le mutazioni puntiformi nella famiglia dei protooncogeni
ras sono state osservate nel 15% - 20% dei tumori umani.
Le mutazioni attivanti di K-ras sono trovate nel 30% degli
adenocarcinomi del polmone, nel 50% dei carcinomi del
colon ed nel 90% dei carcinomi del pancreas.
Le mutazioni di N-ras sono trovate preferenzialmente nei
tumori ematologici, con un'incidenza fino al 25% nelle
leucemie mieloidi acute e nelle sindromi mielodisplastiche.
I membri della famiglia del gene di ras sono
sporadicamente amplificati in vari carcinomi.
Per ricevere il segnale di
attivazione dai recettori dei fattori
di crescita, il ras deve essere
ancorato sotto la membrana
cellulare in prossimità del dominio
intracellulare dei recettori. Tale
ancoraggio è reso possibile
dall’attacco
di
un
gruppo
FARNESILICO che forma dei ponti
tra il ras e la membrana.
Inibitori dell’enzima
farnesiltransferasi
possono inattivare il ras.
Numerosi inibitori FT sono sati sintetizzati:
FTIs sono ben tollerati e la loro tossicità è generalmente reversibile.
Tipifarnib (R115777-Zarnestra) e
Lonafarnib (SCH-66336-Sarasar)
BMS-214662 per via intravenosa.
somministrati per via orale, mentre
Questi composti hanno effetti antitumorali in vivo in animali.
Come singoli chemoterapici hanno effetto nelle leucemie mieloidi.
In tumori solidi l’attività appare modesta, ma FTI probabilmente sono utili
in combinazione con altri agenti citotossici.
ISIS2503 è un oligonucleotide antisense del mRNA di K-ras.
I risultati preliminari non hanno dimostrato risposta obiettiva o alcune
stabilizzazioni di malattia nel cancro polmonare
Drugs that inhibit mutant
KRAS expression or function
Clinics in Chest Medicine , march 2002 Vol 23 n°1:83
Drugs that block specific
downstream signals
active
KRAS
Inactive
KRAS
Inactiv
e
KRAS
GAP
GAP
MAPK/ERK
In vitro è stato dimostrato che
l’inibizione di Raf-1 kinase con
ISIS 5132 induce apoptosi in
cellule tumorali; in vivo, l’attività
antitumorale è confermata in dosi
nel range da 10 a 20 mg/kg
Inibitori di MEK
Il binding dell’
adrenalina al suo
recettore attiva una
G proteina
β
γ
α
La subunità α della G proteina media
l’attivazione dell’ adenilato ciclasi che
porta alla produzione di AMP ciclico
(AMPc)
La subunità catalitica della
PKA fosforila CREB* e
attiva la trascrizione
ATP
GTP
2Pi
Protein chinasi A (PKA)
inattiva
La subunità libera
della PKA migra nel
nucleo
Nucleo
ATP
ADP
P
La subunità
regolatrice della
PKA lega l’AMPc…
…e si dissocia dalla
subunità catalitica
P
Ciclo di attivazione/inibizione delle proteine G
accoppiate a recettori a 7 domini
1. L’ormone lega GPCR, causa un cambio
conformazionale al recettore che è transmesso alla
G-protein.
Ga rilascia GDP e lega GTP (GDP-GTP exchange).
Ciclo di attivazione/inibizione delle proteine G
accoppiate a recettori a 7 domini
2. La sostituzione di GTP con GDP causa un altro
cambio conformazionale in Gα.
Gα-GTP si dissocia dal complesso inibitorio βγ e
può legare e attivare l’Adenilato Ciclasi.
3. L’Adenilato Ciclasi, catalizza da sintesi di cAMP.
4. La Protein Kinase A (cAMP Dependent Protein Kinase)
catalizza il trasferimento di un fosfato da ATP su residui di
serina o treonina di diverse proteine.
Ciclo di attivazione/inibizione delle proteine G
accoppiate a recettori a 7 domini
5. Gα idrolizza GTP in GDP + Pi (GTPasi): Quindi si
riassocia il complesso di Ga al complesso βγ
Adenilato Ciclasi non è più attivato.
6. Le Fosfodiesterasi catalizzano l’idrolisi di cAMP
in AMP.
L’oncogene gsp, che codifica le
subunità delle proteine G, è generato
dalle mutazioni puntiformi. Queste
mutazioni provocano l'attività
costitutiva, portando a stimolo non
regolato dell’adenilato ciclasi.
L’ oncogene gsp è coinvolto nei tumori tiroidei e pituitari,
in cui l’AMP ciclico stimola la proliferazione delle cellule.
Mutazioni simili convertono i geni che codificano per altre
subunità della proteina G in oncogeni in altri tipi di tumori,
compresi i tumori delle surrenali e dell’ovario.
Nonreceptor tyrosine kinases
(32 total in 10 families)
Src family
CSK family
FAK family
JAK family
C-terminal Src kinases
Quale è la funzione cellulare di c-Src?
1. Codificata da un proto-oncogene.
- attività deregolata promuove la proliferazione aberrante
2. Espressa ubiquitariamente.
- più alta in piastrine, neuroni e osteoclasti
- molti funzioni non necessariamente correlate con la
proliferazione
3. Topi src -/- sono sani, nascono e sopravvivono per anni.
- non hanno difetti nelle funzioni neuronali e delle piastrine
- compensato da altre chinasi della famiglia di Src
Espressione delle chinasi della famiglia di Src
Src
Fyn
Yes
Ubiquitario
Ubiquitario
Ubiquitario
Lyn
Hck
Fgr
Blk
Lck
Brain, B-cells, myeloid cells
Myeloid cells
Myeloid cells, B-cells
B-cells
T-cells, NK cells, brain
4. src/fyn/yes tripli knockout l’embrione muore a metà-gestazione.
- cellule hanno difetti nella motilità ma non nella proliferazione
- difetti nella motilità revertiti dalla re-espressione di c-Src
5. c-Src and v-Src si localizzano prevalentemente nelle adesioni
focali.
- consistente con il suo ruolo nella motilità cellulare
Src contiene tre dominii presenti anche in altre
proteine
Lega sequenze ricche in polyproline
Dominio
fosforilante
myristate
palmitate
Lega proteine fosforilate in Tyr
Site whose phosphorylation by Csk
DECREASES Kinase activity
La fosforilazione della tirosina terminale
crea
una
interazione
intramolecolare
inibitoria
Src è di norma inattivo a causa della
inibizione intramolecolare
La struttura
tridimensionale di
Src
Xu et al. Nature. 1997 385:595-602
L'attività chinasi di questa proteina è inattivata normalmente da
fosforilazione del residuo della tirosina in posizione 527, che è
posta a sei residui dal C-terminale.
La tirosina in posizione 527 è
spesso mancante o alterata nelle
oncoproteine Src, a causa della delezione di 18 amminoacidi
del suo C-terminale, di conseguenza la chinasi è
costituzionalmente attiva; cioè non richiede l'attivazione
da una fosfatasi.
La fosforilazione delle proteine target da parte delle
oncoproteine aberranti Src contribuisce alla proliferazione
anormale di molti tipi cellulari.
Activation/VEGF
! One of the signaling pathways that c-Src is involved in is the VEGF (Vascular Endothelial
Growth Factor)
Involment of c-Src in the VEGF pathway
SABiosciences, ProteinLounge.com
Le placche di adesione, per mezzo delle quali le cellule interagiscono
con ECM da una parte e con le actine citoscheletriche dall’altra,
sono siti di intensa fosforilazione in tirosina.
Green = actin
Red = pTyrosine
Yellow = costain
(focal adhesions)
Adesioni
Focali
Lamina basale!
Adesioni Focali:
Regioni specializzate della
membrana plasmatica
formate ai siti dove le cellule
aderenti legano il substrato
ECM
Filamenti
Actina !
Adesioni
Focali!
ECM
- Siti di clasterizzazione delle integrine, recettori per le glicoproteine
adesive dell’ECM
- Siti di ancoraggio dei filamenti di actina >> invasione e motilità cellulare
costituiti da due subunità α e β
(esistono 16 α e 8 β per un totale di 22
distinte integrine)
mediano l’adesione tra la cellula e le
proteine dell’ECM, quali fibronectina,
laminina, collagene e, in alcuni casi,
anche con “contro-recettori” di altre
cellule quali
“intracellular cell-adhesion
molecule-1” (ICAM-1) e
“vascular cell-adhesion
molecule-1” (VCAM-1)
Fisiologia Cellulare e Molecolare AA 2009-2010 - Parte 3°
ECM
Struttura delle Integrine
ligandi:
Legame con molecole della ECM
es. fibronettina, collageno,
vitronectina,
α β
ligandi solubili es. fibrinogeno
membrana plasmatica
citoplasma
Legame al citoscheletro e molecole
di segnalamento
proteine che legano l’actina
es. vinculina, a-actinina, talina,
Proteine che legano i filamenti
intermedi es. plakoglobina
Molte molecole di segnalamento es.
tirosine chinasi
L’ottimale segnalamento dell’integrina richiede sia
occupazione dell’integrina sia la clasterizzazione dell’integrina
CITOPLASMA
Tensin FAK
RGD
Occupazione
Actin stress fibres
Talin a-actinin
Vinculin Tensin FAK
RGD RGD RGD RGD
Clasterezzazione
Occupazione e
clasterizzazione
a-actinin
actin
Tensin
Pax
Small GTPases FAK
-Rho,Rac,cdc42 G
src
SS
Cation-bindingM2+
D
site
G SP
R
G
CITOPLASMA
Tensin
Pax
FAK
G src
SS
M2+
D
G SP
R
G
ECM glycoprotein
eg fibronectin
A seconda della taglia e della
localizzazione le strutture
possono essere contatti focali,
adesioni focali, o adesioni
fibrillari.
I segnali trasdotti attraverso
i complessi focali e le adesioni
focali sono implicati in una
serie di processi cellulari
Le Integrine interagiscono con l’actina, con proteine della trasduzione e a recettori 1TM
modulazione di
canali del Ca2+
Clusterizzazione
delle integrine
riorganizzazione
citoscheletrica
Ca2+
Tirosin
chinasi
FAK
(focal adhesion kinase)
Serin treonin
chinasi
Src
Rho
Proteine G
monomeriche
Modulazione delle
adesioni e delle
giunzioni
ciclina D - cki
caspasi - p53
proliferazione
sopravvivenza
Complesso Adesione Focale
COMPONENTI STRUTTURALI
Talina è la struttura base
delle adesioni focali,
lega actina, vinculina, e FAK.
a-actinina è una
proteina di collegamento
che può legare anche la
vinculina.
Vinculina lega actina e tensina.
Segnalamento attraverso Integrina
α β
FAK
Grb-2
PI3-K
MAPK
Proliferation
Survival
Akt
Struttura di FAK (Focal Adhesion Kinase)
tirosina chinasi non-recettore (come la famiglia Src
–nuotante nel citosol
Nck
Crk
Cas Sos
Integrin ?
Csk
talin
paxillin
Crk
FAT Region
Amino Terminal Domain
Kinase Domain
Y925
Y397
Src
Grb-7
Carboxy-terminal Domain
Grb-2
PI3-K
Sos
FAT Region = Focal Adhesion Targeting Region
Integrine, FAK, e MAPK
Intracellulare
Matrice Extracellulare
fibronectina
β
integrina!
α
fibronectina
1!
P
src
Paxillina
fak
MEMBRANA
2!
3!
P!
4!
Src
Intracellulare
5!
Organizazione
del citoscheletro
(rac, rho, ras)
Segnalamento via ECM
1 Aggregazione integrine
2 Autofosforilazione di fak
3 Associazione fak/src
4
5!
Nucleo
5
src fosforila fak
risposte mitogenetiche
5 AND...
FAK
Dopo il legame al complesso attivato
dell'integrina, FAK autofosforila la sua
tirosina
c-Src è una tirosin chinasi non
recettore citosolica solitamente
inattiva per la configurazione
auto-impacchettata
Questo
fosfotirosina
permette che FAK
leghi i dominii SH2
di Src o Fyn
fosforilandoli
FAK legandosi attiva c-Src aprendola
C-Src fosforila FAK di nuovo!
c-Src chinasi
FAK chinase
solo legame
SH2 domain of Grb2
Attivazione di
JNK/NF-kB
Sopravvivenza
Cellulare
(proteina adattatrice recrua
RasGEF SOS alla membrana)
attivano
Ras-MAPK
p85 subunità di PI-3K
attivazione
PKB/AKT
Sopravvivenza e
Propagazione
.... e la adesione tra cellule e
con la ECM
• giunzioni strette
• placche di adesione
• citoscheletro
Src substrates
Src coordina la motilità
cellulare e la adesione
3-dimensional structure of Src
Active site
Scientists have determined the precise 3-dimensional structure of Src
Active site
SU6656
Another Src inhibitor is in Phase I/II trials
for metastatic pancreatic cancer
Active site
AZD0530
Attivazione di JNK/NF-kB
JNK è un modulatore delle funzioni delle cellule epiteliali che è
implicato in risposte opposte.
Quando è attivato dalle integrine porta alla progressione del
ciclo cellulare attraverso l’espressione di geni AP1.
Le integrine attivano anche il fattore di trascrizione NF-kB, che
promuove la sopravvivenza e l’angiogenesi.
L'overespressione di integrine è un
forte sostegno per le cellule del tumore
Integrina α5β3
fortemente espressa
sul fronte invasivo del melanoma maligno
e dei vasi sanguigni angiogenici.
Espresso a debolmente
in nevi e vasi sanguigni quiescenti
Integrina α6β4
fortemente espressa
NON espressa
durante la progressione dei carcinomi tiroidei
nelle cellule tiroidee benigne
Integrina α5β1
Ha un azione soppressiva del tumore.
La sua espressione è persa in molti tumori
Integrine e Cancro
• Il profilo delle integrine cambia nella
progressione di molti tumori solidi
• Spesso, specifiche integrine sono perse
• Transfezione per ripristinare l’espressione
dell’integrina decrementa la tumorigenicità
Espressione di αvβ6 in epitelio orale normale e
carcinoma delle cellule squamose
x33
epitelio normale
x33
carcinoma Squamoso
FAK Knockout mice
• Embrioni letali
– Embrioni hanno severi difetti morfogenici
– Il numero delle adesioni focali è
INCREMENTATO
• Non difettano nella formazione delle adesioni
focali, come fibroblasti FAK-/- fanno
eccezionalmente larghe adesioni focali
• Il difetto sembra essere nella dissoluzione
dell’adesione focale, critica per la migrazione
cellulare
FAK e Cancro
• In molti tumori solidi, l’espressione di FAK
è aumentata
• FAK può essere un marker di tumori
invasivi e metastatici
• Aumentate sopravvivenza e migrazione,
funzioni dell’aumentata espressione di
FAK fa di esso un attraente target per lo
sviluppo di piccole molecole inibitorie
come agenti antineoplastici
FAK e SOPRAVVIVENZA
• L’ingranaggio di FAK dall’integrina stimola un
pathway della sopravvivenza
• Il distacco delle cellule epiteliali dalla ECM
provoca un tipo di apoptosi. ANOIKIS
• La trasduzione del segnale procede attaverso
altri enzimi di segnalamento che promuovono la
sopravvivenza: FAK-PI3Kinasi-Akt-Bad-antiapoptosi
• Questa funzione può essere la principale di FAK
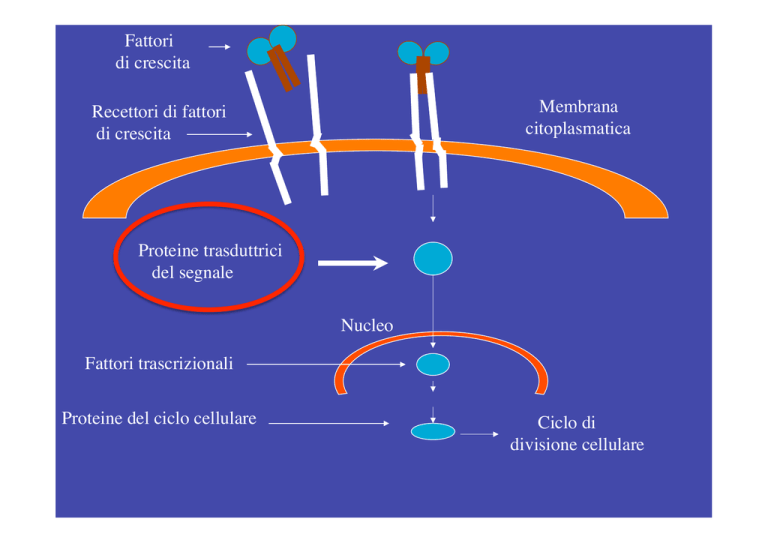
Fattori
di crescita
Membrana
citoplasmatica
Recettori di fattori
di crescita
Proteine trasduttrici
del segnale
Nucleo
Fattori trascrizionali
Proteine del ciclo cellulare
Ciclo di divisione cellulare
Fattori di Trascrizione
Fattori di Trascrizione
Legano specifiche sequenze (enhancer) del
DNA
Sono up-regulatori della trascrizione
Rendono la regione del promotore
accessibile alla RNA polimerasi II
Molti fattori di trascrizione sono i bersagli
finali di specifiche vie di trasduzione
La croma(na ha un ruolo fondamentale nella regolazione della espressione genica e la capacità degli istoni di legare il DNA è regolata da enzimi intranucleari che possono apportare modificazioni post-­‐traduzionali sopra=u=o ai residui di lisina e arginina pos( nel dominio N-­‐terminale delle proteine istoniche MODIFICAZIONI POST-­‐TRADUZIONALI DEGLI ISTONI ace$lazione, fosforilazione, me$lazione, ubiqui$nazione Zhang Y , Reinberg D Genes Dev. 2001;15:2343-2360
Me$lazione degli istoni Nei vertebrati la metilazione interessa solamente la Citosina sul dinucleotide CpG:
l’enzima citosina metiltransferasi aggiunge un gruppo metile al C5 della citosina: il
risultato è la 5-metilcitosina.
•
Le sequenze CpG sono sotto-rappresentate nel genoma ma abbondanti nelle regioni
promotrici dei geni
•
In cellule non embrionali, 80% dei CpG sono metilati, ad eccezione delle isole CpG
dei promotori
•
Dnmt1 ha particolare affinità per le sequenze emi-metilate: tende quindi a metilare il
nuovo filamento che si è formato su uno stampo metilato-> mantenimento del
pattern di metilazione
Generalmente la metilazione degli istoni si associa a repressione della espressione genica,
tuttavia ci sono esempi in cui la metilazione si associa a attivazione della espressione genica
(metilazione lella lisina 4 dell’istone 3, il coattivatore CARM1 è una metiltransferasi)
PROCESSI DI ACETILAZIONE E DEACETILAZIONE SONO IMPORTANTI NELLA REGOLAZIONE DELLA ATTIVITA’ DELLA CROMATINA ESPRESSIONE GENICA E’ GENERALMENTE ASSOCIATA CON ACETILAZIONE L’ace(lazione dei residui di lisina al terminale NH degli istoni rimuove le cariche posi(ve riducendo quindi l’affinità degli istoni per il DNA: per questo l’ace(lazione degli istoni facilita i processi trascrizionali, al contrario, la deace(lazione reprime la trascrizione Ace(lazione e deace(lazione del residuo lisinico ACETIL TRANSFERASI ISTONICHE: histone acetyltransferases (HATs) DEACETILASI ISTONICHE: histone deacetylases (denoted by HDs or HDACs)
Acetilazione degli Istoni
•
Diminuiscono la condensazione Il macchinario di trascrizione puo’ accedere al DNA
meno condensato
Repressori: deacetilazione degli Istoni
•
•
Alcuni repressori attirano delle deacetilasi
Prevengono l’accesso del macchinario di
trascrizione al DNA
– Acetilazione/
Deacetilazione
•
•
Suberoyl anilide bishydroxamide
SAHA
Corepressori
CoR, BcoR e CtBP.
Uno dei meccanismi che
favorisce la carcinogenesi
è la repressione dei geni
onco-soppressori.
•
•
p21
p300/CBP
TAF II 250
Classe I: HDAC1, 2, 3 e 8.
Classe II: HDAC 4, 5, 6, 7, 9 e 10.
! Fattori di Trascrizione
!
!
I fattori di trascrizione appartengono a ciascuna delle diverse classi basate su specifici tipi di domini di
legame
Molti contengono un α-elica, che si inserisce nel solco maggiore del DNA
! Legame fra αα e DNA via mediante:
! Forze di Van der Waals (hydrophobic)
! legami Ionici
! Legami idrogeno
! Motivi comuni ai fattori di trascrizione
! Zinc
finger:
Zn coordinato a due cisteine e due istidine
! Leucine
zipper
Cerniere di leucina
! Helix-loop-helix
Elica –giro-elica
2 eliche separate da un loop
1. Attivazione trascrizionale (Fos)
2. Fosforilazione (Fos, Jun, CREB, STAT)
3. Defosforilazione (Jun, CREB)
4. Scambio di Partner (Myc, Jun)
5. Rilascio da inibitori (NFkB, SMADs)
6. Legame del ligando (‘recettori’ nucleari)
i fattori di trascrizione AP-1 (activator protein 1) sono substrati della
trasduzione
Famiglia FOS
C-fos
Fra 1
Fra 2
Fos B
Famiglia JUN
C-jun
Jun B
Jun D
PI3K
P
PDK
PKA
P
PKC
MAPK
(ERK/JNK)
fos-jun leucine-zipper domains
P
P
jun-jun
AP-1
p90rsk
PLCβ/γ
GF
PLCβ/γ
SIERO
VOCs
Ca2+
STAT
Smad
MAPK
PKC
TCF/Elk1
SIE
-350
SRF
SRE
cAMP
CaMK IV
Fos/jun
AP1-RE
-300
AC
PKA
CREB
Ca/CRE
C-fos
TATA
-60
Geni della risposta tardiva
Time course dell’espressione dei geni
della risposta precoce e di quelli tardivi in
cellule in G0 dopo l’aggiunta di mitogeni
SRE, serum response element
SRF, proteina fattore responsivo al siero
TCF, fattore del complesso ternario
AP1-RE, sito di legame della proteina attivatrice
Ca/CRE, l’elemento responsivo al calcio e al cAMP
Geni della risposta precoce
Attivazione Transcrizionale e post-translazionale di
AP-1.
Attivazione di AP-1 è stimolata da un complesso
network di vie segnalamento che partono da segnali
esterni e MAPKs:
ERK, p38, JNK. Le MAPKs attivano vari fattori di trascrizione: TCF, (Fattore del complesso terziario), MEF2C, enhancer fattore 2C dei miociti (ATF2) fattore 2 attivante la trascrizione e JUN.
FOS e JUN
AP1
La Fosforilazione Post-translatzionae regola
l’attività di AP-1, che include la sua
transattivazione, la capacità di legame al DNA e la
stabilità dei componenti di AP-1. CKII, casein kinase II; GSK-3, glycogeno sinthase
kinase-3; RSK2, ribosomal S6 kinase 2; SRE,
serum-response element.
The four MEF2 transcription factors (MEF2A to
MEF2D)
regulate
calcium-dependent
gene
expression in muscle cells have a role in the
differentiation of cardiac and skeletal muscle. The expression of muscle-specific genes is accomplished through MEF2-responsive
elements (MEF2 sites).
Post-translational modifications of jun
JUN è phosphorylated on
Ser63 and Ser73 da JNK,
increasing its stability and
transactivation potential.
Phosphorylations of Thr91
and Thr93 are required for
DNA
binding
and
activation
of
its
transcriptional activity. JUN ubiquitylation required
phosphorylation at Thr239 by
glycogen synthase kinase 3
(GSK3), after phosphorylation on Ser243.
The effect of GSK3
can be antagonized by
the
dephosphorylation of
stability
Ser243 by
calcineurin. ubiquitina ligase the leucine zipper
domain
ERK and PI3K–Akt signalling cascades results in JUN stabilization.
JUN can be sumoylated on Lys257 and Lys229, which leads to a reduced transcriptional activity. ERK induces the acetylation of the lysine residues in the JUN DNA binding region, thereby increasing
JUN transcriptional activity. Oncogenic activation - what alterations?
b ZIP
b ZIP
TAD
v-Jun
a common principle that underlies oncogenic
mutations - to escape regulation by kinases or other
modifying enzymes, leading to constitutive activity.
The protein encoded by the avian sarcoma virus 17 oncogene v-Jun shows increased
transforming activity compared with c-Jun, its normal cellular counterpart. v-Jun differs from c-Jun in three important ways that might explain its transforming
potential: (1) deletion of the delta domain - Jnk docking?, (2) single amino-acid
substitutions that change a phosphorylation sites and (3) site that is recognized by the
redox factor Ref1
Oncogenic activation
Contrariamente a c-Jun, sia JunB che JunD mancano di
attività trasformante. Anzi, le proteine JunB e JunD
hanno effetti anti-oncogénici; infatti le cellule della
linea mieloide caratterizzati dalla mancanza di junB, a
conseguenza del silenziamento epigenetico, hanno
un'apoptosi ridotta, che correla con l'aumento
d'espressione di due geni anti-apoptotici, BCL-2 e
BCL-XL. 1- L’attività di AP-1 è stimolata dal trattamento con il promotore tumorale TPA e da fattori di crescita.
c-FOS può cambiare il pattern di metilazione del DNA regolando il gene DNA 5-methylcytosine
transferase, portando alla downregolazione di geni oncosoppressori. c-JUN–c-FOS contribuisce al
tumore primario cooperando con i prodotti di altri oncogeni, come RAS.
2- JUNB and JUND essendo downregulati non consentono la formazione di dimeri oncogenici. I
complessi c-JUN/AP-1 impediscono l’apoptosi mediata da p53. 3- I complessi c-FOS/AP-1 inducono espressione di geni: angiogenesis e invasività. Nei tessuti adiacenti AP-1 stimola la secrezione di fattori paracrini, cge influenzano la crescita del
tumore
i fattori di trascrizione responsivi al cAMP:
CREB/CREB
CREB/CREM
CREB/fos
CRE
Several genes + Alternative splicing generates
several variants
Distinct gene products, such as:
CREB
CREBP1
CREM
ATF1-4
Alternative splicing in CREM
generates isoforms acting both as activators and repressors
Signalling through cAMP and PKA to CREB
AC
α
β
γ
cAMP
PKA
Cytoplasm
Dissociation C
C
C
The cyclic AMP response element-binding
protein (CREB) regulate a wide range of
biological functions, such as growth factordependent cell proliferation and survival,
glucose homeostasis, spermatogenesis,
circadian rhythms and memory. Nuclear translocation
C
Phosphorylation
P
CBP
P
CREB
Nucleus
P
Target gene activation
The crucial event in the activation of CREB is the phosphorylation of Ser133 in kinase-inducible
domain (KID) and Ser133 promotes CREB to recruit transcriptional co-activators that induce
transcription of a variety of intermediate early response genes.
protein kinase A (PKA), protein kinase C (PKC), casein kinases, calmodulin kinases (CaMKs), glycogen
synthase kinase-3, p34cdc2, p70s6k] can either increase or decrease the activity of CREB. Dephosphorylation of Ser133 is important for the inactivation of CREB. Both protein phosphatase 1
(PP-1) and PP-2A may be involved in the dephosphorylation of CREB
i diversi segnalamenti intracellulari che mettono capo a CREB
MAPK-activated protein
kinase 2 Ser/Thr protein
kinase regulated through
direct phosphorylation by p38
MAP kinase
cyclins, Bcl-2 family members, Egr-1
(Early growth response protein 1)
In mammals, four RSK genes (RSK1-RSK4)
and two MSK genes (MSK1 and MSK2) have
been identified. RSK and MSK are
ubiquitously expressed and many cell types
express several members of each family.
CREB is overexpressed and constitutively phosphorylated in a number of forms of human
cancer, including acute myeloid leukemia (AML) and non–small cell lung cancer, and
appears to play a direct role in disease pathogenesis and prognosis. Turning off the response- the ICER strategy
AC
α
β
γ
cAMP
PKA
C
C
Dissociation
Cytoplasm
C
Nuclear translocation
C
ICER
Phosphorylation
P
Competition
Inactive
heterodimers
CBP
Repressor TURN OFF
P
P
CREB
Nucleus
Target gene activation
The inducible cyclic AMP early repressor (ICER) is a group of proteins produced from the CREM/ICER, which
contains only a DNA binding domain, functions as a repressor of transcription of several CRE
A pro-apoptotic role of ICER in neurons could be of special importance, especially because CREB appears to
have antiapoptotic activity in cultured neurons deprived of trophic support
ETS (Ets1, Ets2, PntP2); YAN; ELG; PEA3 (Erm, ER81); ERF; TCF (Elk1, Sap1a, Sap1b, Sap2)
Proteine TCF (Elk e Sap-) si legano ad un dimero SRF:SRF (serum-response-factor)
e controllano RRE (Ras-Responsive-Elements)
TCF = Ternary Complex Factor
85
In molte cellule la transizione proliferazione
differenziazione
vede la down-regolazione di Myc
MXD1
MXD2
MaX-Interactor 1
MXD3
MXD4
eccessivi livelli di Myc portano
alla formazione di tumori
Le proteine MXD1-4 (dette in passato MAD) sono
repressori trascrizionali correlati a Myc, e antagonizzano
l’attività oncogenica di Myc
reclutano il corepressore Sin3 e il complesso contenente
HDAC
Le proteine Leucine zipper Max, Mnt, Miz1 e Mlx interagiscono con
MXD e Myc
MXD1
Myc-Associated factor X
MaX-Dimerization protein 1
c-Myc è un gene della risposta
precoce regolato da fattori della
comunicazione
Transactivation
Myc
MB1
MB2
Myc
mRNA
Max
Max
DNA Binding
BR HLH
LZ
BR HLH
LZ
Max, Mnt, Miz1 e Mlx interagiscono con MXD e Myc
La dimerizzazione regola MYC
1 ora
MYC richiede Max per legare il DNA
GF
Mentre Myc è GF-inducibile,
Max è costitutivo
myc indotto dai
fattori di crescita
viene neutralizzato
da Miz1
Myc
repressione
Max
Max
i fattori di crescita
inducono l’espressione
di myc portando
all’attivazione di geni
bersaglio
repressione
Max
MXD1
Sin 3
repressione
Max
Miz1
repressione
Myc
Max
attivazione
Max
MXD2
Sin 3
Le funzioni attivanti e inibenti di Myc promuovono la proliferazione, l’apoptosi e la
tumorigenesi e sopprimono la differenziazione
MYC-MAX si lega alla sequenza E-box e recluta
HATs tipo GcN5, TiP60 e transformation/
transcription domain-associated protein (TRRAP),
con successiva acetilazione vicino il
transcriptional start site (Tss).
Però, MAX può dimerizzare con membri della
famiglia Mxd tipo MAX dimerization protein 1
(MXD1), MAX interactor 1 (MXi1), MXD3, MXD4 e
MAX binding protein (MNT). Questi dimeri
competono con MYC-MAX e reclutano HDACs
con successiva repressione
MYC–MAX possono essere reclutati dal fattore
di trascrizione MiZ1 e si portano sul initiator
element (inr) di vari geni reclutando la DNA
(cytosine-5)-methyltransferase 3α (DNMT3A)
reprimendo la trascizione di geni dipendenti da
MiZ1
Indipendentemente da MAX, MYC forma
complessi con il transcription factor IIIB (TFIIIB)
stimolando la trascrizione di geni dipendenti
dalla RNA polymerase III (transfer RNA (tRNA)) e
5S ribosomal RNA (5s rRNA) reclutando TRRAP
e GcN5
A, acetylation; M, methylation.
p27
p27
Uno dei geni target di myc-max target è cdc25. Cdc25 è una fosfatasi che rimuove un
fosfato dal complesso ciclina-cdk che
promuove la transizione G2-M. il “network”
regolatorio di MYC
Myc è indotto da MAPK tramite
fosforilazione di Ets
MAPK
Myc
Ets
E2F
Myc ha una emivita di 30 min; la sua stabilità è controllata dalla fosforilazione su Ser-62 e Thr-58.
La fosforilazione su Ser-62 stabilizza Myc, mentre quella su Thr-58, successiva alla fosforilazione Ser-62, promuove la
degradazione
GF
PI3K
ERK
_
PKB/Akt
Myc
GSK-3
Ser 62
P
Myc
P
STABILE
Myc
P
Myc
INSTABILE
UBIQUITIN
cis trans
Myc
PIN-1
P
P
FWB7
T58-MYC è target della
ubiquitina ligasi FWB7
Thr 58
P
PP2A
trans-MYC è substrato di PP2A
P
Myc
la fosforilazione operata da
GSK-3 promuove il legame
di prolyl isomerasi (PIN-1)
che cambia la
configurazione di MYC da
prolina cis a prolina trans
Myc ha una emivita di 30 min; il suo turnover è controllato da ERK, PKB/AktGSK-3 e da PP2A
PP2A consists of a dimeric core enzyme composed of the structural
A and catalytic C subunits, and a regulatory B subunit. Protein CIP2A also known as cancerous inhibitor of PP2A
PKB/Akt
_
GSK-3β
instabile
stabile
ERK
La fosforilazione su Ser-62 da parte di ERK stabilizza
Myc, mentre quella su Thr-58, successiva alla
fosforilazione Ser-62, e operata da GSK-3, promuove il
legame di prolyl isomerasi (PIN-1) che cambia la
configurazione di MYC da prolina cis a prolina trans ;
trans-MYC è substrato di PP2A; T58-MYC è target della
ubiquitina ligasi (degradazione)
Nelle cellule normali l’espressione di CIP2A è bassa
e la defosforilazione operata da PP2A è consentita.
In alcuni tumori i livelli di CIP2A sono elevati
portando al mascheramento di MYC e alla sua
stabilizzazione
CIP2A inhibits PP2A tumor suppressor activity in human malignancies as prevent c-Myc proteolytic degradation. In accordance with the oncogenic role of CIP2A, overexpression promotes Ras-elicited cell growth and transforms
immortalized human cells Myc richiede Max per legare il DNA
Myc–Max legati a Miz1
reprimono la
trascrizione
Myc–Max
attivano la
trascrizione
Le funzioni attivanti e inibenti di Myc promuovono la proliferazione,
l’apoptosi e la tumorigenesi e sopprimono la differenziazione
Mad–Max sono repressori trascrizionali; quando sovraespressi hanno attività
anti-proliferativa e anti-apoptotica, influenzano negativamente la differenziazione
e hanno capacità oncosoppressoria
Oltre a Max, Mad dimerizza con Mlx.
Come i dimeri Mad–Max, Mad–Mlx
reprimono la trascrizione
c-myc
L’attivazione di myc avviene attraverso molti
meccanismi:
amplificazione genica –
double-minutes e espansione di regioni di cromosomi
– translocazioni che pone myc sotto il controllo di
altri promotori.
Amplificazioni of c-myc
-Breast carcinoma
-neuroblastoma (gene N-myc)
- cancro polmone a piccole cell. (gene L-myc)
TRASLOCAZIONI del gene c-myc si verificano nel linfoma di
Burkitt, un tumore delle cellule B.
La traslocazione dal
cromosoma 8q24 al
cromosoma 14q32 porta cmyc vicino al gene che
codifica per la catena
pesante delle
immunoglobuline. In questi
casi la traslocazione porta ad
una continua stimolazione del
gene c-myc da parte degli
enhancers posti vicino ai geni
per le immunoglobuline.
Nel 10-15% delle leucemie linfoblastiche acute delle
cellule T (T-ALL), la trascrizione di c-myc è sotto il
controllo degli elementi regolatori all'interno del
locus della catena alfa del recettore delle cellule T.
Vβ
Vα
γ ε
ζ ζ
ε δ
Cβ Cα
fyn
Zap 70
lck